Абрикос – ценнейшая культура, возделываемая человеком более 6000 лет, ее ареал охватывает Китай, горные районы Средней Азии, доходя на западе до Передней Азии и Закавказья [1, 2]. Были описаны 3 основных центра происхождения абрикоса: Китайский, Центрально-Азиатский и Ближневосточный [3]. Культивируемый абрикос, Prunus armeniaca L. (Rosaceae, подсемейство Prunoidae), является третьим по важности видом косточковых культур и имеет довольно маленький геном (2n = 16). Эта культура распространяется по всему миру, но большая часть сосредоточена в районе Средиземного моря, что составляет более 55 % мирового производства. Биологические и хозяйственно ценные свойства абрикоса вызывают большой интерес к изучению этой культуры в различных странах мира. Абрикос сочетает в себе такие биологические особенности, как интенсивный рост, скороплодность, долголетие, высокую продуктивность. Плоды абрикоса содержат большое количество витаминов, благодаря чему широко используются не только как диетический, но и лекарственный продукт. Азербайджан богат различными сортами фруктов, среди которых особое место занимают абрикосы. Основные территории, пригодные для разведения абрикоса, сосредоточены в Нахичеване, Тертере и Агдаше. История выращивания абрикоса насчитывает три тысячи лет. В результате длительной народной селекции в Азербайджане созданы такие сорта, как алыча-ерик, аг новретсе, гырмызы новретсе, хагверди, агчанабат, аг теберзе, балйарым, тохум шемси, гара теберзе, сары теберзе, абуталиби, хосров шахи.
Наиболее актуальным направлением в селекции различных растений является применение молекулярных маркеров для паспортизации, изучения полиморфизма ДНК, генетических взаимоотношений и выявления генов, контролирующих хозяйственно ценные признаки [4]. Сочетание менее трудоемкого морфологического анализа с методом молекулярного маркирования приводит к более надежным выводам для оценки генетического разнообразиям плодовых растений [5–7]. Для характеристик зародышевой плазмы абрикоса во всем мире используются ряд молекулярных маркеров, включая ISSR, AFLP, SSR и др. [8–10]. Наиболее широкое применение получил метод SSR-маркирования. Высокая надежность, мультиаллельная природа, кодоминантный характер наследования и хромосомоспецифичность микросателлитных маркеров делают их удобными для генетического анализа и изучения межвидовых генетических связей [4, 11]. Несмотря на большое количество публикаций по генетическим исследованиям абрикоса данное направление все еще остается актуальным [12–14]. Отметим, что абрикос с использованием современных молекулярных методов мало изучался в Азербайджане.
Цель исследования: изучение генетического разнообразия и генетических связей 61 образцов абрикоса Азербайджанского происхождения с использованием 7 SSR маркеров.
Материалы и методы исследования
Молекулярные исследования проводили на 61 сорте абрикоса из четырех регионов Азербайджана (Нахичевань, Тертер, Агдаш и Астара). Названия и происхождения изученных генотипов приведены в табл. 1.
Выделение ДНК и ПЦР
Геномную ДНК выделяли из свежсобранных листьев СТАБ методом [15]. Концентрацию и степень чистоты молекулы ДНК определяли с помощью спектрофотометра NanoDrop2000c. ПЦР-смесь (20 мкл) включала 1,5 мкл 10× ПЦР буфера, 1,5 мкл смеси dNTP (10 мM), 1 мкл MgCl2 (50 мM), 0,35 мкл 10 µM праймера, 0,3 мкл фермента Taq полимеразы (5 U/мкл), 0,20 мкл флуоресцентной метки (FAM, NED, PET и VIC) и 3 мкл выделенной ДНК (50 нг/мкл).
Анализ данных
Для каждого SSR локуса было рассчитано общее число аллелей (Na). Параметры генетического разнообразия, включающие частоту встречаемости основных аллелей, наблюдаемую (HO) и ожидаемую (HE) гетерозиготность, а также величину информационного полиморфизма (PIC) были статистически анализированы с применением программного обеспечения PowerMarker, версия V3.025 [16]. Для молекулярного анализа были использованы 7 микросателлитных маркеров: aprigms18, ssrPaCITA16, ssrPaCITA19, ssrPaCITA4, UDAp-404, ssrPaCITA21, pchgms2. Кластерный анализ проводили при помощи программы DARwin 6.0 [17].
Результаты исследования и их обсуждение
Генетический полиморфизм. В любой селекционной программе важно иметь возможность выявить уникальные ДНК профили для сортовой идентификации, определения генетического разнообразия, выявления родительских форм и изучения таксономического родства. Использование SSR маркеров обеспечивает возможность решения этих задач в генетике и селекции плодовых культур рода Prunus. Известен целый ряд публикаций, связанных с изучением генетического полиморфизма абрикоса на основе микросателлитных повторов [18–20].
Для молекулярного анализа 61 образца абрикоса из четырех регионов Азербайджана был использован SSR-метод (табл. 2). В результате исследования по 7 микросателлитным локусам было синтезировано 59 аллелей. Число аллелей на локус находилось в пределах 6 (для маркеров ssrPaCITA16 и ssrPaCITA21) – 12 (для маркера UDAp-404) и в среднем составило 8,5.
Таблица 1
Названия и географическое происхождение образцов абрикоса
|
№ |
Образец |
Место сбора |
№ |
Образец |
Место сбора |
|
1 |
Зейнеби |
Агдаш |
32 |
Ордубад эрики |
Tертер |
|
2 |
Май Натиг |
Агдаш |
33 |
Аг эрик |
Tертер |
|
3 |
Аг эрик Гюльнар |
Агдаш |
34 |
Бадам эрик |
Tертер |
|
4 |
Йени форма |
Нахичевань |
35 |
Шемси |
Нахичевань |
|
5 |
Чыр Зеферан |
Нахичевань |
36 |
Бадам эрик |
Геранбой |
|
6 |
Чыр эрик |
Нахичевань |
37 |
Агча Набат |
Нахичевань |
|
7 |
Неизвестный |
Нахичевань |
38 |
Гейча Набат |
Нахичевань |
|
8 |
Майчичейи |
Нахичевань |
39 |
Хагверди |
Нахичевань |
|
9 |
Балйарым |
Нахичевань |
40 |
Иран сорту |
Нахичевань |
|
10 |
Хампа |
Нахичевань |
41 |
Ордубад Шерефи |
Нахичевань |
|
11 |
Йени форма |
Нахичевань |
42 |
Хейдери |
Нахичевань |
|
12 |
Чыр Нахичевань |
Нахичевань |
43 |
Ордубад чыры |
Нахичевань |
|
13 |
Йай Шерефи |
Нахичевань |
44 |
Форма |
Нахичевань |
|
14 |
Шалах |
Нахичевань |
45 |
Ирандан гелме |
Нахичевань |
|
15 |
Теберзе |
Нахичевань |
46 |
Ордубад Набати |
Нахичевань |
|
16 |
Тохум Шемси |
Нахичевань |
47 |
Йени форма |
Нахичевань |
|
17 |
Гечйетишен |
Нахичевань |
48 |
Шалах |
Нахичевань |
|
18 |
Бадами |
Нахичевань |
49 |
Алча эрик |
Нахичевань |
|
19 |
Хелена (Американский сорт) |
Нахичевань |
50 |
Абу Телеби |
Нахичевань |
|
20 |
Мехмани |
Нахичевань |
51 |
Теберзе |
Нахичевань |
|
21 |
Хагверди |
Нахичевань |
52 |
Аг Эрик Эльчин |
Агдаш |
|
22 |
Аг Набати |
Нахичевань |
53 |
Май Геранбой |
Агдаш |
|
23 |
Кюрдеши |
Нахичевань |
54 |
Майовка |
Агдаш |
|
24 |
Талеби |
Нахичевань |
55 |
Бадами |
Агдаш |
|
25 |
Туркийе сорту |
Нахичевань |
56 |
Шалах |
Агдаш |
|
26 |
Аг бадами |
Нахичевань |
57 |
Гырмызыйанаг |
Тертер |
|
27 |
Агчанабад |
Нахичевань |
58 |
Иреван эрик Шалах |
Тертер |
|
28 |
Лимон эрик |
Нахичевань |
59 |
Майовка |
Тертер |
|
29 |
Форма |
Нахичевань |
60 |
Лимон эрик |
Нахичевань |
|
30 |
Аг эрик (позднеспелый) |
Геранбой |
61 |
Аскерабат |
Нахичевань |
|
31 |
Аг эрик (скороспелый) |
Геранбой |
В селекционном процессе любых плодовых культур, в том числе абрикоса, важно выявить уникальные генетические профили ДНК для идентификации и определения генетических взаимоотношений [21–23]. В наших исследованиях общее число уникальных аллелей составило 12 (диапазон 1–5). Уникальные аллели были обнаружены в локусе pchgms2 у генотипов Зейнеби, Май Натиг, Чыр Зеферан, Елена и Ордубад Шерефи (хромосома 7, пять аллелей), в локусе UDAp-404 у генотипов Зейнеби, Чыр Зеферан и Елена (хромосома 4, три аллеля), в локусе ssrPaCITA19 у генотипов Зейнеби, Май Натиг и Елена (хромосома 2, три аллеля), в локусе ssrPaCITA4 у генотипа Майовка (Агдаш) (хромосома 3, один аллель). Максимальное число уникальных аллелей было выявлено праймером pchgms2. Наличие уникальной комбинации аллелей показано для шести сортов, содержащих от одного до трех таких аллелей. У образцов Зейнеби и Елена зафиксированы уникальные аллели в трех локусах (ssrPaCITA19, UDAp-404, pchgms2). Наличие уникальных аллелей, обнаруженных у некоторых образцов, может быть связано с тем, что эти сорта были обогащены зародышевой плазмой различного происхождения или же с мутацией в микросателлитной последовательности, которая дает начало новому аллелю. Таким образом, маркеры pchgms2, UDAp-404 и ssrPaCITA19 являются наиболее эффективными и могут быть рекомендованы для идентификации образцов абрикоса.
Нами были рассчитаны частоты встречаемости выявленных аллелей и индекс информативности (PIC) для каждого маркера (табл. 2).
Частота основных аллелей варьировала от 0,221 (ssrPaCITA4) до 0,418 (ssrPaCITA16) и в среднем равнялась 0,342. Полученные значения PIC находились в диапазоне 0,54–0,8, что указывает на информативность большинства использованных SSR маркеров. Наименьший показатель PIC выявлен для локуса ssrPaCITA21, наибольший – для ssrPaCITA4.
Таблица 2
Основные показатели генетического разнообразия у 61-го образца абрикоса
|
Локус |
Группа сцепления |
Общее число аллелей |
Частота основного аллеля |
HO |
HE |
PIC |
|
aprigms18 |
1 |
9 |
0,3689 |
0,98 |
0,78 |
0,75 |
|
ssrPaCITA16 |
2 |
6 |
0,4180 |
0,87 |
0,69 |
0,66 |
|
ssrPaCITA19 |
2 |
8 |
0,3033 |
0,98 |
0,75 |
0,71 |
|
ssrPaCITA4 |
3 |
7 |
0,2213 |
0,94 |
0,82 |
0,8 |
|
UDAp-404 |
4 |
12 |
0,2869 |
0,6 |
0,76 |
0,73 |
|
ssrPaCITA21 |
5 |
6 |
0,4016 |
0,35 |
0,61 |
0,54 |
|
pchgms2 |
7 |
11 |
0,3934 |
1 |
0,66 |
0,61 |
|
Среднее значение |
8,4 |
0,3420 |
0,82 |
0,72 |
0,68 |
|
|
Всего |
59 |
Примечание. HO – величина наблюдаемой гетерозиготности, HE – величина ожидаемой гетерозиготности, PIC – величина информационного полиморфизма.
Показатели наблюдаемой (HO) и ожидаемой (HE) гетерозиготности находились в пределах 0,35–1 (в среднем 0,82) и 0,61–0,82 (в среднем 0,72) соответственно. Установлена корреляция между наблюдаемым и ожидаемым уровнем гетерозиготности. Наибольшие значения ожидаемой гетерозиготности были выявлены праймерами ssrPaCITA4 (HE -0,82), aprigms18 (HE -0,78), UDAp-404 (HE -0,76) и ssrPaCITA19 (HE -0,75). Полученные нами результаты показали, что изученные зародышевые плазмы абрикоса имеют высокий уровень генетического разнообразия и согласуются с данными других авторов. Так, исследование Жебентяева и соавторов [24] о генетическом разнообразии основных абрикосов культивируемых в Европе, Северной Америке, Южной Африке и Австралии показали, что ожидаемая гетерозиготность в среднем составила 0,645. Бургибу и другие [25] исследуя генетическое разнообразие абрикосов культивируемых в Алжире, Марокко и Тунисе выявили ожидаемую гетерозиготность равной 0,593, которая была ниже, чем у абрикосов, выращиваемых в Китае. Чжан и соавторы [26], проанализировали 94 сортов абрикоса в Китае с использованием SSR маркеров и показали высокий уровень генетического разнообразия, где средняя ожидаемая гетерозиготность была равна 0,792.
Методом кластерного анализа генотипы сгруппировались в 7 различных кластеров, где индекс генетического расстояния (ИГР) варьировал от 0 до 1 (рисунок). Наибольшее количество генотипов объединились в кластере 7, который подразделяется на 2 субкластера. Второй и пятый кластер включают в себя по 11 генотипов, четвертый и шестой – по 9, а наименьшим числом образцов представлен кластер 3. Между некоторыми сортами выявлено нулевое значение коэффициента генетического сходства (ИГР = 0). Генотип под номером 46 сформировал отдельный кластер, что говорит о наибольшей отдаленности этого сорта. Максимальное генетическое расстояние установлено между генотипами под номерами 2 и 58, 46 и 19, 24 и 29, 54 и 28, 51 и 28, где ИГР был равен 1, что дает возможность их применения в различных селекционных программах с целью обогащения генетического разнообразия абрикоса. В изучаемой коллекции собраны отечественные образцы абрикоса из четырех регионов Азербайджана. Анализируя дендрограмму, можно отметить группировку в зависимости от места сбора изученных сортов. Во всех кластерах превалируют образцы из Нахичевани, что обусловлено наибольшим размером выборки. А второй кластер представлен исключительно образцами из вышеуказанного региона. Сорта 1, 2, 52 и 54 из Агдаша сгруппировались в шестом, сорта 33, 57 и 58 из Тертера в третьем кластере, что может свидетельствовать об их генетическом сходстве. Существует ряд работ, направленных на анализ генетических родственных связей сортов абрикоса [27, 28]. Проведение таких исследований позволяет оценить существенные генетические различия между группами сортов.
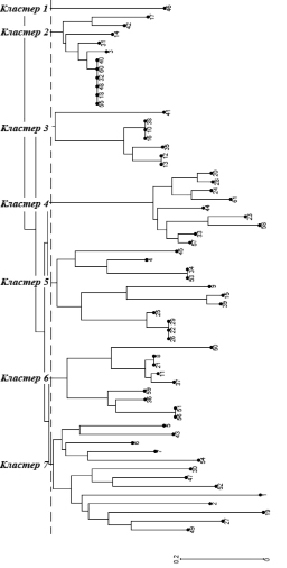
Дендрограмма, построенная на основе индекса генетического расстояния
Заключение
Таким образом, был изучен полиморфизм семи микросателлитных локусов и выявлено богатое генетическое разнообразие сортов изученной коллекции абрикоса, что подтверждает эффективность выбранных SSR маркеров. Наиболее информативными оказались праймеры ssrPaCITA4, aprigms18, UDAp-404 и ssrPaCITA19, которые отличились высокими показателями гетерозиготности и наибольшими значениями PIC. Кроме того, в изученной выборке были обнаружены сорта с уникальными аллелями, выявленными праймерами pchgms2, UDAp-404 и ssrPaCITA19. Указанные праймеры могут быть использованы для идентификации и паспортизации сортов абрикоса в дальнейших – селекционных программах.



