Основная часть золота, ежегодно добываемого во всем мире, в частности в США, Китае, Австралии, России, странах Европы и Африки, извлекается с помощью цианистого выщелачивания. Используемые цианиды являются чрезвычайно токсичными химическими соединениями, способными связываться с металлами и инактивировать ферменты дыхательной цепи [1]. Помимо золотодобывающей промышленности, источниками цианидов служат гальваническое, газовое, лакокрасочное и металлургическое производство, газификация угля. В связи с этим на сегодняшний день актуальна проблема их утилизации [2].
Соединения, содержащие циано-группу (–C≡N), в природе присутствуют в различных формах, но токсичность их зависит от способности образовывать свободный цианид (CN–). Чаще они встречаются в виде металл-цианидных комплексов. Например, в виде легко диссоциируемых комплексов с никелем, медью и цинком или более устойчивых соединений с железом и кобальтом. Другие важные производные цианида представлены цианатом (OCN–), образующимся при окислении цианида, тиоцианатом (SCN–) – продуктом взаимодействия между свободной формой цианида и присутствующей в рудах восстановленной серой, нитрилами или циангидринами, органическими формами цианида. Пути разложения цианидов различны. Они основываются на реакциях окисления, восстановления, замещения, присоединения и гидролиза.
Несмотря на токсичность этих соединений, многие организмы, в том числе бактерии, грибы, водоросли, растения и некоторые животные, способны как синтезировать цианиды, так и ассимилировать. Обширная группа микроорганизмов использует в своем метаболизме CN– и SCN– в качестве источников энергии и серы и/или азота, что делает ее потенциальным объектом для использования в биологической детоксикации сточных вод и растворов металлургических производств. Такая способность обнаружена у бактерий родов Arthrobacter, Pseudomonas (P. pseudoalcaligenes, P. fluorescens, P. stutzeri, P. putida, P. diminuta), Bacillus (B. pumilus), Acinetobacter (A. johnsonii), Halomonas (H. meridians), Klebsiella (K. pneumoniae), Ralstonia, Acidovorax, Escherichia, Methylobacterium, Thiobacillus (T. thioparus), а также у Alcaligenes xylosooxidans subsp. denitrificans, Paracoccus thiocyanatus, Thioalkalivibrio thiocyanoxidans, Achromobacter xylosoxidans, Thiohalophilus thiocyanoxidans и др. При этом некоторые микроорганизмы, помимо цианидов, попутно окисляют тиосульфаты, тетратионаты, сульфиды и элементную серу, расширяя области возможного промышленного применения [2, 3].
Цель работы: исследовать таксономический состав сообщества микроорганизмов, выделенного из кека фильтрации после переработки сульфидной золотосодержащей руды и принимающего участие в ассимиляции и диссимиляции цианидов и извлечении золота, с помощью высокопроизводительного секвенирования.
Материалы и методы исследования
В работе использовали микробное сообщество, выделенное в НИГТЦ ДВО РАН из кека фильтрации, полученного после цианирования золотосодержащей руды месторождения Аметистовое (Камчатский край). Технологическая усредненная проба кека, светло-коричневого цвета, без запаха, влажностью 21,5 % и сохраненная при температуре от –10 до +30 °С, имела содержание золота 0,39 г/т, серебра – 5,6 г/т.
Выделение микробной биомассы осуществляли в периодическом режиме в биореакторе с механическим перемешиванием путем добавления к кеку (225 г) дистиллированной воды (1800 мл) в соотношении Т:Ж 1:8. Культивирование происходило при средней температуре 27 °С в течение 8 дней. На момент окончания процесса количество микроорганизмов в растворе, определяемое прямым подсчетом на микроскопе с фазово-контрастной насадкой «МИКРОМЕД 3 вар. 3-20» (Россия, Китай), насчитывало 3,6·107 кл/мл.
Таксономический состав выделенной биомассы исследовали с помощью высокопроизводительного секвенирования маркерного гена 16S рРНК, выполненного в Федеральном исследовательском центре «Фундаментальные основы биотехнологии» Российской академии наук (Москва) по договору о НИР [4].
В ходе молекулярно-биологических исследований ДНК выделяли следующим стандартным методом. Образец ресуспендировали в 200 мкл лизирующего буфера (0,15 M NaCl, 0,1 M Na2EDTA (pH 8,0)), содержащего 15 мг/мл лизоцима. После чего инкубировали на водяной бане при 37 °C в течение 60 мин, тщательно встряхивая лизат каждые 10 мин. Затем вносили аналогичное количество 200 мкл буфера (0,1 M NaCl, 0,5 M Tris-HCl (pH 8,0)) и додецилсульфат натрия до конечной концентрации 0,5 %. Дальнейшее разрушение клеток осуществляли с помощью гомогенизатора FastPrep® 24 (MP Biomedicals, США) в соответствии с инструкциями производителя. После гомогенизации проводили обработку протеиназой К, которую добавляли до конечной концентрации 100 мкг/мл, после чего лизат инкубировали в течение 40 мин на водяной бане при 50 °C. Эффективность лизиса определяли подсчетом клеток в световом микроскопе с фазово-контрастным устройством. Затем к лизату добавляли равный объем смеси фенол-хлороформ (фенол был насыщен 0,1 M Tris-HCl (pH 8,0); хлороформ представлял собой смесь хлороформа и изоамилового спирта в соотношении 24:1). Полученную смесь аккуратно перемешивали в течение 10–20 мин. Смесь центрифугировали при 12000 x g в течение 8 мин. Затем отбирали супернатант, который еще 2 раза аналогичным образом обрабатывали хлороформом. К получившемуся объему супернатанта добавляли 0,1 часть 3 М ацетат натрия (рН 5,2) и 3 части 96 % спирта. Для осаждения ДНК данную смесь инкубировали при –20 °C в течение ночи. Затем ДНК осаждали центрифугированием при 12000 x g в течение 20 мин. Осадок последовательно промывали и центрифугировали при 12000 x g в течение 8 мин сначала в 70 % спирте, затем в 96 %, после чего супернатант удаляли. Полученный осадок тщательно высушивали и растворяли в 30–50 мкл ТЕ буфера (10 мM Tris-HCl, 1 мM EDTA (pH 8,0)). Получившийся раствор ДНК очищали от примесей РНК путем инкубации с РНКазой А (конечная концентрация 0,2 мг/мл) в течение 2 ч при 37 °C. Также ДНК очищали с помощью набора крупных фрагментов ДНК Wizard® DNA Clean-Up System (Promega, США). Качественная и количественная оценка полученных препаратов ДНК производилась на спектрофотометре DropSense-96® (Trinean, Бельгия). После лизиса буфер с раствором ДНК хранили при –20 °С в течение приблизительно 70 сут [4].
Библиотеки для секвенирования были приготовлены в соответствии с ранее описанным протоколом. В качестве универсальных использовали праймеры Pro341F(5’-CCTACGGGNBGCASCAG-3’) и Pro805R (5’-GACTACNVGGGTATCTAATCC-3’), которые позволяют амплифицировать вариабельные участки генов 16S рРНК V3 и V4. Для дальнейшего секвенирования полученные ампликоны разделяли с помощью электрофореза в агарозном геле. Вырезанные из геля ампликоны очищали с помощью набора для очистки ДНК из геля и реакционных смесей Cleanup Standard (Евроген, Россия) [4].
Секвенирование проводили при помощи набора реагентов, обеспечивающего длину прочтения 300 нуклеотидов с каждого конца ампликона. Первичная обработка (фильтрация и демультиплексирование) полученных прочтений производилось при помощи ПО CLC Genomics Workbench 7.5 (Qiagen, США). Полученные данные были обработаны с помощью онлайн-сервиса SILVAngs (https://www.arb-silva.de/ngs/). Всего было проанализировано 59392 фрагмента средней длиной 486 нуклеотида [4].
Результаты исследования и их обсуждение
Оценка выделенных таксономических единиц в ходе метагеномного секвенирования 16S рРНК показала присутствие 181 известного рода и неклассифицированных групп микроорганизмов. Таксономическое разнообразие на уровне доменов было представлено практически только бактериями. Археи насчитывали лишь тысячные доли процента (рисунок, таблица). До вида идентифицировать микроорганизмы не удалось.
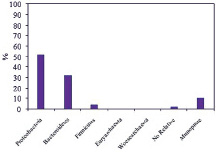
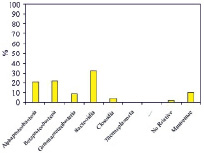
а) б)
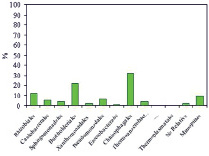
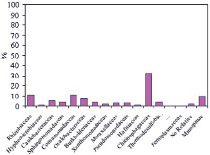
в) г)
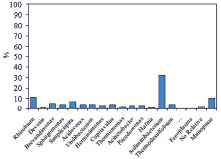
д)
Таксономический состав сообщества микроорганизмов, выделенного из кека фильтрации при переработке золотосодержащей руды месторождения Аметистовое, на уровне филов (a), классов (б), порядков (в), семейств (г) и родов (д)
Таксономический состав представителей микробного сообщества, выделенного из кека фильтрации при переработке золотосодержащей руды месторождения Аметистовое
|
Домен |
Филум |
Класс |
Порядок |
Семейство |
Род |
Количество, % |
|
Bacteria |
Proteobacteria |
Alphaproteobacteria |
Rhizobiales |
Rhizobiaceae |
Rhizobium |
11 |
|
Hyphomicrobiaceae |
Devosia |
1 |
||||
|
Caulobacterales |
Caulobacteraceae |
Brevundimonas |
5 |
|||
|
Sphingomonadales |
Sphingomonadaceae |
Sphingomonas |
4 |
|||
|
Betaproteobacteria |
Burkholderiales |
Comamonadaceae |
Simplicispira |
7 |
||
|
Acidovorax |
4 |
|||||
|
Oxalobacteraceae |
Undibacterium |
4 |
||||
|
Herminiimonas |
3 |
|||||
|
Burkholderiaceae |
Cupriavidus |
4 |
||||
|
Gammaproteobacteria |
Xanthomonadales |
Xanthomonadaceae |
Thermomonas |
2 |
||
|
Pseudomonadales |
Moraxellaceae |
Acinetobacter |
3 |
|||
|
Pseudomonadaceae |
Pseudomonas |
3 |
||||
|
Enterobacterales |
Hafniaceae |
Hafnia |
1 |
|||
|
Bacteroidetes |
Bacteroidetes |
Chitinophagales |
Chitinophagaceae |
Sediminibacterium |
32 |
|
|
Firmicutes |
Clostridia |
Thermoanaerobacterales |
Thermodesulfobiaceae |
Thermodesulfobium |
4 |
|
|
Archaea |
Woesearchaeota (DHVEG-6) |
− |
− |
− |
− |
0,003 |
|
Euryarchaeota |
Thermoplasmata |
Thermoplasmatales |
Ferroplasmaceae |
Ferroplasma |
0,003 |
|
|
No Relative |
No Relative |
No Relative |
No Relative |
No Relative |
No Relative |
2 |
|
Доля остальных минорных членов сообщества |
9,994 |
|||||
Дальнейшему анализу подвергали доминирующие таксономические единицы, доля которых составляла ≥ 1 %. На уровне филумов бактерии характеризовались присутствием Proteobacteria, Bacteroidetes и Firmicutes. Причем на протеобактерии приходилось более 50 %. Таксономический состав включал 5 классов, представленных главным образом Bacteroidia, Betaproteobacteria и Alphaproteobacteria, 9 порядков с доминированием Chitinophagales, Rhizobiales и Burkholderiales, 13 семейств с явным превалированием Chitinophagaceae и 15 родов, среди которых преобладал Sediminibacterium.
Бактерии рода Sediminibacterium представлены строго аэробными грамотрицательными палочками, способными к гетеротрофному росту и выделяемыми из почв и различных водных объектов (сточные воды, пресная вода и др.) [5]. Информации о непосредственной причастности данных бактерий к синтезу или разрушению цианидов в литературе не имеется. Однако в результате того, что микроорганизмы были выделены сравнительно недавно и слабо изучены, о действительной их роли сложно судить.
Микроорганизмы, которые имеют отношение к ассимиляции или разложению циандиов, в анализируемом сообществе были представлены в меньшем количестве. Так, бактерии рода Rhizobium – второго (11 %) рода после Sediminibacterium, ранее выделяемые из перерабатывающих никельсодержащие цианиды биореакторов, способны к ассимиляции большого количества цианида [6]. Есть сведения о том, что идентифицированные в настоящей работе представители родов Sphingomonas (например, S. paucimobilis) и Acidovorax способны к деструкции цианида и тиоцианата. Немаловажная роль Acinetobacter подтверждается данными о способности разрушать широкий спектр цианидсодержащих соединений, включающих комплексные соединения цианидов с металлами, свободные цианиды и простые органические нитрилы [7].
В исследуемом сообществе выявлен род Hafnia. Известно, что у бактерии (H. alvei) данного рода обнаружена роданаза – внутриклеточный фермент, катализирующий образование тиоцианата из тиосульфата и цианида, что подтверждает ее причастность к деструкции последнего. Также совсем недавно стало извечтно об участии бактерий рода Thermomonas, выделенных из сточных вод и систем биоочистки отходов после коксования, в биодеструкции цианида [8]. Стоит отметить, что род Devosia был выявлен в составе альгобактериального сообщества, разлагающего тиоцианат [9].
Обнаруженные в сообществе кека фильтрации представители рода Pseudomonas играют двоякую роль, так как участвуют как в синтезе, так и в разрушении цианида [10, 11].
Некоторые присутствующие микроорганизмы могут быть связаны не столько с метаболизмом цианидсодержащих соединений, сколько с разрушением сульфидов и участвовать в окислительно-восстановительных реакциях с металлами. Например, представитель рода Herminiimonas (H. arsenitoxidans) способен к окислению арсенита [12]. Бактерия рода Cupriavidus (C. metallidurans), обладающая устойчивостью в отношении ряда катионов металлов (меди, цинка, серебра, кадмия, свинца и золота), доминирует в формирующихся на частицах золота биопленках. Это, вероятно, связано с ее участием в биоминерализации металла [13].
Отдельно необходимо отметить присутствие в сообществе анаэробных хемолитотрофных бактерий рода Thermodesulfobium, относящихся к сульфатредукторам, которые были недавно выделены из кислых термальных источников Камчатки [14]. Поскольку сульфатредукторы обладают способностью образовывать тиосульфат, нельзя исключать их потенциальное участие в деструкции цианидов.
Неизвестна роль остальных бактерий родов Undibacterium, Brevundimonas и Simplicispira, входящих в состав проанализированного сообщества, в деструкции или синтезе цианидов. Однако в связи с выделением данных микроорганизмов из различных природных источников, таких как почвы, питьевые и промышленные сточные воды и активный ил [15], их присутствие может быть связано с широкой областью распространения.
Среди выявленных доминирующих бактерий большая часть принадлежала к известным охарактеризованным родам. Микроорганизмы были представлены в основном грамотрицательными мезофильными аэробными палочками, для которых характерен гетеротрофный способ питания, за исключением рода Thermodesulfobium – автотрофных сульфатредуцирующих бактерий.
Заключение
На основании проведенного высокопроизводительного секвенирования 16S рРНК сообщества микроорганизмов, выделенного из кека фильтрации после цианирования золотосодержащей руды месторождения Аметистовое, анализ таксономического состава показал обширную группу хорошо известных и некультивируемых микроорганизмов, представленных главным образом грамотрицательными мезофильными аэробными бактериями, для которых характерен гетеротрофный способ питания.
В составе доминирующих представителей сообщества были обнаружены как типичные бактерии, способные к синтезу и деструкции цианида и его производных (роды Rhizobium, Sediminibacterium, Sphingomonas, Acidovorax, Acinetobacter, Hafnia, Pseudomonas и др.), так и микроорганизмы, принимающие участие в окислительно-восстановительных реакциях трансформации серо- и металлсодержащих соединений (роды Herminiimonas и Cupriavidus). Превалирующим по количеству (32 %) являлся род Sediminibacterium, роль которого в синтезе или разложении цианидов не известна. Однако он, как и остальные представители анализируемого сообщества, встречается в природных и техногенных объектах, что не исключает потенциальной роли в ассимиляции и диссимиляции циансодержащих соединений.
Авторы благодарят к.б.н., заведующего лабораторией хемолитотрофных микроорганизмов ФИЦ «Фундаментальные основы биотехнологии» РАН А.Г. Булаева за методическую помощь и консультации, а также сотрудников НИГТЦ ДВО РАН А.В. Киореску, В.О. Мусихина и А.С. Хомченкову за техническую помощь в проведении работ по выделению микроорганизмов.
Библиографическая ссылка
Пашкевич Р.И., Хайнасова Т.С. ИЗУЧЕНИЕ СОСТАВА СООБЩЕСТВА МИКРООРГАНИЗМОВ, ВЫДЕЛЕННОГО ИЗ КЕКА ФИЛЬТРАЦИИ ПОСЛЕ ПЕРЕРАБОТКИ СУЛЬФИДНОЙ ЗОЛОТОСОДЕРЖАЩЕЙ РУДЫ // Успехи современного естествознания. 2019. № 12-2. С. 301-307;URL: https://natural-sciences.ru/ru/article/view?id=37305 (дата обращения: 07.03.2026).
DOI: https://doi.org/10.17513/use.37305



